近日,我组与上海交通大学鲁洪中副教授合作,在酵母系统生物学研究中取得新进展。研究团队通过整合分析全球1807株酿酒酵母菌株的基因组与生态位数据,构建了高覆盖度的酿酒酵母泛基因组及菌株特异性代谢模型,系统解析了酵母通过基因组精简与代谢重编程适应多样化环境的分子机制。
酿酒酵母在烘焙、酿酒等领域具有悠久的应用历史,在自然与人工环境中均展现出强大的适应性进化能力。尽管不同进化分支的菌株在遗传和表型上存在显著多样性,但其在代谢水平上的差异尚不清晰。目前,全球科研与工业界主要依赖少数实验室菌株(如S288c,CEN.PK)作为出发菌株,这极大地限制了对酵母宝贵自然多样性资源的开发与利用。因此,如何筛选最优菌株以构建服务于医药、能源、化工等领域的酵母细胞工厂,成为亟待解决的问题。
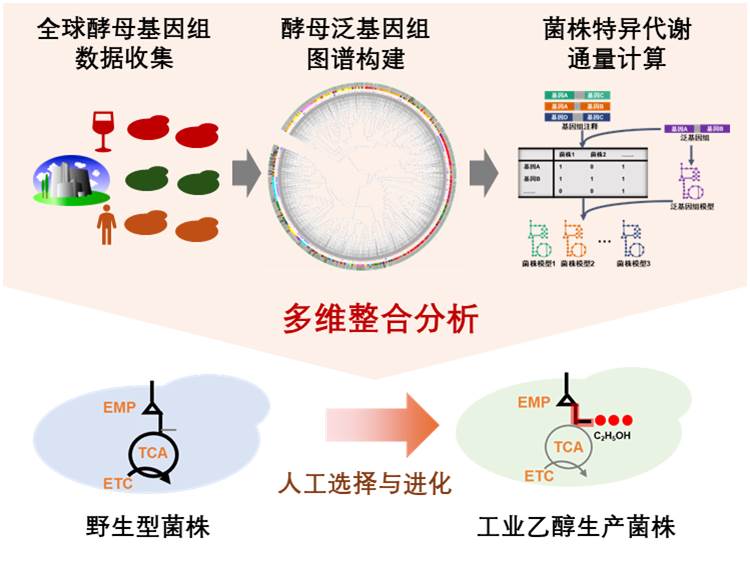
本研究创建了高质量酵母泛基因组与菌株特异性代谢模型数字资源库。基于此,团队创新性地建立了“泛基因组-代谢模型-多组学整合”的多维度分析流程,为从系统层面理解微生物的环境适应性及代谢特性提供了新理论框架。以工业乙醇生产菌株为例,团队从基因、转录及代谢通量三个维度证实,强化糖酵解下游途径与乙醇的高效合成密切相关,该发现为后续乙醇及其衍生物细胞工厂的理性设计提供了重要线索。
该工作以“Yeast adapts to diverse ecological niches driven by genomics and metabolic reprogramming”为题,发表在《美国国家科学院院刊》(Proceedings of the National Academy of Sciences)上。我组博士研究生王浩宇为论文第一作者,丹麦生物创新研究院Jens Nielsen教授为文章提供了重要指导。以上工作得到国家重点研发计划、国家自然科学基金等项目的资助。(文/图 王浩宇 校对/王倩)
文章链接:https://doi.org/10.1073/pnas.2502044122

